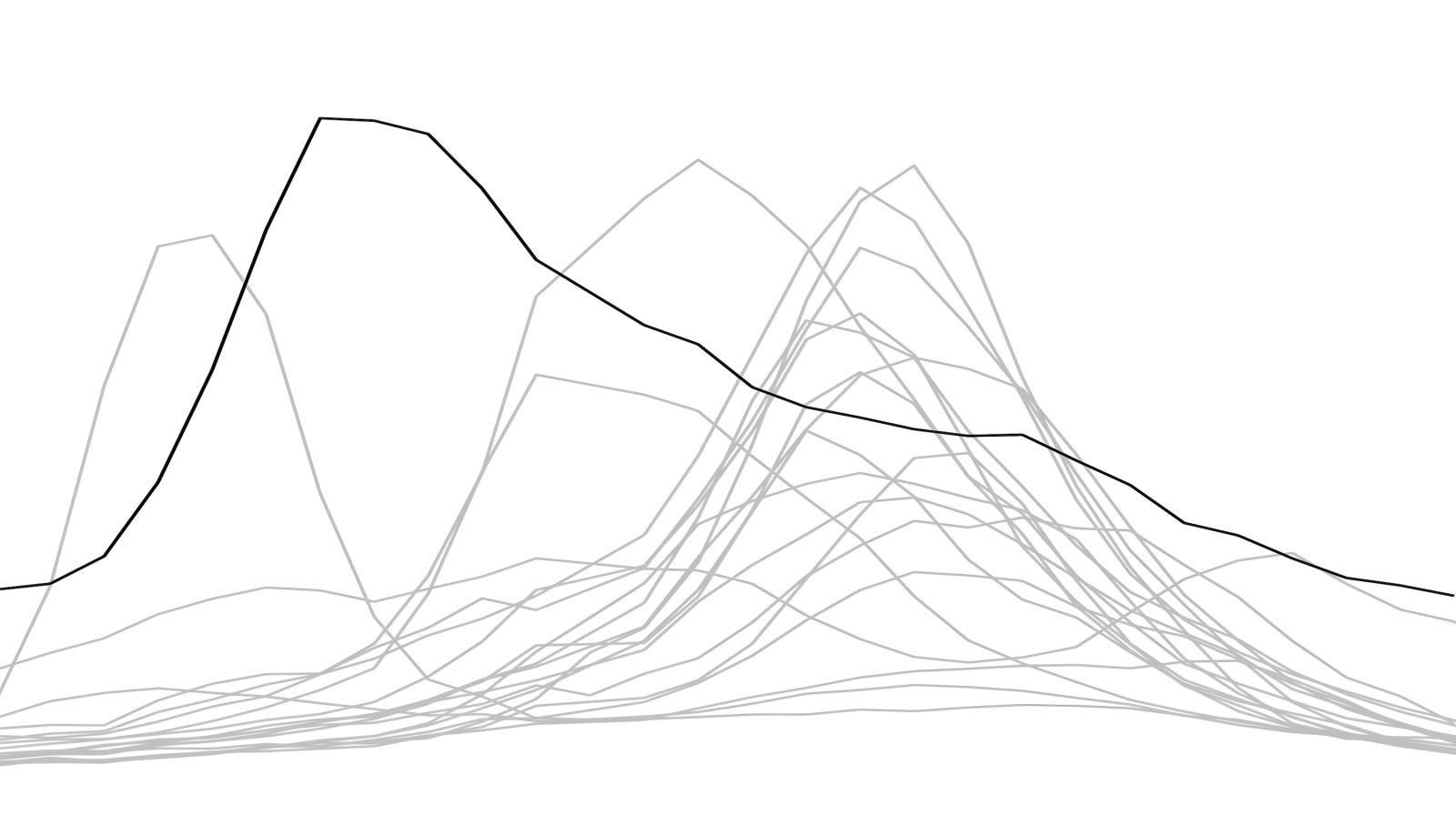
Il progetto pilota del portale open source di sorveglianza epidemiologica cerca di rispondere alla sfida del rilascio e mantenimento dei dati open, sviluppando una rete sempre più numerosa di esperti. L'obiettivo è condividere e valorizzare i dati in un'ottica One Health, basandosi sulla collaborazione intersettoriale per supportare il rilevamento, il monitoraggio e la valutazione del rischio per le malattie infettive emergenti e riemergenti in Europa. Nella figura l'andamento delle ILI (Influenza like illness) dal portale InfluNet di Francesco Branda e Luca Cozzuto.
Quando la popolazione mondiale era piccola e scarsamente distribuita, le malattie infettive erano letali ma locali. La globalizzazione e i continui spostamenti delle persone hanno trasformato virus e batteri in assassini di massa. In meno di vent'anni, si sono verificate tre gravi epidemie che hanno interessato la popolazione umana: la SARS nel 2003, l’influenza aviaria H1N1 nel 2009 e la sindrome respiratoria del Medio Oriente (MERS) nel 2012. E ancora, Ebola, Zika, HIV/AIDS, West-Nile fino ad arrivare alla sindrome respiratoria acuta grave Coronavirus-2 (SARS-CoV-2). La migliore difesa contro un nuovo agente patogeno mortale è la soppressione aggressiva dei primi focolai, che richiede una loro rapida individuazione. La pandemia causata dal SARS-CoV-2 ha riportato all’attualità l’importanza dell’apertura, della condivisione e dell’utilizzo dei cosiddetti real-world data per generare prove scientifiche che possano consentire ai decisori politici di agire in modo informato e considerare in modo adeguato, nel breve e medio periodo, i compromessi fra le conseguenze sanitarie e gli effetti sociali ed economici delle misure adottate.
Diverse tipologie di dati sono fondamentali per la gestione di un focolaio epidemico: (i) i dati genomici dei patogeni servono per identificare l'agente causale di un'infezione, tracciare le mutazioni e studiare le reti di trasmissione e diffusione geografica di una malattia; (ii) i dati clinici sono utili per comprendere la gravità della malattia, sviluppare la definizione dei casi clinici, valutare e monitorare gli esiti degli interventi farmaceutici; (iv) i dati sierologici sono importanti per caratterizzare l'immunità di un individuo e le sue risposte anticorpali; (v) i dati epidemiologici, che vanno dal conteggio aggregato dei casi a informazioni dettagliate sul tracciamento dei contatti, sono fondamentali per caratterizzare parametri di base come il numero di riproduzione (R0), cioè il numero medio di persone che sono contagiate da ciascuna persona infetta, e le distribuzioni temporali chiave (dall'insorgenza dei sintomi all'ospedalizzazione).
Negli ultimi anni, l'uso dei dati aperti, comunemente chiamati con il termine inglese Open Data, è stato fondamentale per: (i) condurre un’analisi della situazione in tempo reale, rintracciare i contatti e fare diagnosi precoci e tempestive per un contenimento efficace del contagio; (ii) facilitare il coordinamento e la collaborazione tra i governi nazionali e locali; (iii) garantire la fiducia del pubblico nei confronti dei decisori politici attraverso una maggiore trasparenza; (iv) contrastare la disinformazione; (v) identificare e affrontare le vulnerabilità e le esigenze particolari di determinate fasce della popolazione raccogliendo dati disaggregati; e (vi) sostenere una gestione efficace delle forniture e delle richieste di attrezzature mediche.
Le sfide legate all'analisi dei dati riguardano l'intero ciclo di vita di un'epidemia. Nelle fasi iniziali, è necessario acquisire conoscenze fondamentali sulle caratteristiche epidemiologiche di una nuova infezione, dal potenziale di trasmissione alla storia naturale. Ciò richiede l’uso di test diagnostici e il sequenziamento genomico, oltre a una rapida valutazione dell'impatto clinico e una condivisione aperta dei primi risultati. Con la crescita dei focolai, è necessario prevedere le dinamiche della malattia, stimare il carico potenziale e valutare l'efficacia degli interventi per ritardare o appiattire la curva epidemica. Nelle fasi successive, l'attenzione si rivolge alla stima dell'efficacia del vaccino e al monitoraggio dei focolai e delle dinamiche evolutive. Ciò può includere l'analisi di una serie di dati a lungo termine, come gli studi di coorte che possono fornire approfondimenti sui processi epidemici nell'arco di mesi o addirittura anni.
L’emergenza coronavirus ha rivelato le debolezze nel modo in cui i paesi raccolgono, gestiscono e usano le informazioni, ma ha anche posto le basi per la prevenzione e il controllo delle future pandemie, utilizzando il cosiddetto modello delle 3M: Monitoring, cioè monitorare l'evoluzione dell'epidemia e valutare l'efficacia delle contromisure adottate; Modelling, cioè modellare e prevedere la diffusione dell'epidemia; e Managing, cioè prendere decisioni tempestive per gestire, mitigare e sopprimere il contagio. La recente pandemia può essere un'opportunità per rinnovare l'attenzione sul miglioramento della qualità, della tempestività e della completezza dei dati sanitari prodotti dai governi, rafforzando le pratiche di gestione delle informazioni esistenti.
Il successo delle iniziative sui dati aperti richiede lo sviluppo di processi in grado di garantire che gli standard di de-identificazione delle informazioni siano rispettati, assicurando la pubblicazione di metadati completi, impegnando risorse per la manutenzione continua e rendendo la condivisione dei dati aperti una funzione governativa di routine. Tuttavia, la pubblicazione di dati liberamente accessibili sul web non è sufficiente. Sono necessarie politiche proattive per stimolare un insieme di attori reciprocamente interdipendenti con ruoli e funzioni diverse, come dimostrato dall’uso creativo dei dati aperti su COVID-19 da parte di giornalisti, ricercatori e sviluppatori software.
Un portale di sorveglianza
Il continuo rilascio e mantenimento di dati aperti di qualità è una sfida cui il progetto pilota del nostro portale open source di sorveglianza epidemiologica (risultato anche degli studi condotti durante il mio dottorato di ricerca) cerca di rispondere, ponendosi l’obiettivo di diffondere e promuovere la cultura Open Data e Open Science nell’ambito della ricerca sanitaria. Il progetto ha lo scopo di sviluppare una rete sempre più numerosa di professionisti e ricercatori per affrontare le sfide della condivisione e della valorizzazione dei dati in un quadro di One Health basato sulla collaborazione intersettoriale per supportare il rilevamento, il monitoraggio e la valutazione del rischio per le malattie infettive emergenti e riemergenti in Europa.
Con un’interfaccia utente user-friendly e dal design minimalista, è possibile accedere direttamente ai dati grezzi, documentarsi sulla tipologia di dataset, visualizzare in anteprima i campi di interesse, e infine scaricare in locale i file necessari per le proprie analisi. Gli argomenti trattati - vaiolo delle scimmie (o Mpox), febbre del Nilo (o West Nile Virus), influenza stagionale e aviaria, epatite acuta a eziologia sconosciuta in età pediatrica - hanno dimostrato la flessibilità del sistema, fornendo dati epidemiologici affidabili, pronti per l’uso e ben definiti a seconda del periodo e del relativo interesse della comunità scientifica.
Sulla base dei dati raccolti dalle fonti ufficiali di monitoraggio sanitario (Organizzazione Mondiale della Sanità, Centro europeo per la prevenzione e il controllo delle malattie e Istituto Superiore di Sanità) e lo sforzo collaborativo di virologi, medici, statistici, data scientist, ingegneri informatici e bioinformatici è stato possibile: (i) contribuire a comprendere le origini e le dinamiche di trasmissione della recente epidemia di Ebola in Uganda; (ii) stimare parametri epidemiologici chiave nelle prime fasi di Mpox; (iii) implementare modelli predittivi per seguire e prevedere la diffusione di COVID-19 in tempo reale; (iv) sviluppare un’applicazione web che raccoglie, confronta e analizza le informazioni epidemiologiche e virologiche dell'influenza stagionale. In quest’ultimo caso, la condivisione dei dati è stata fondamentale anche per promuovere iniziative di comunicazione e sensibilizzazione, come nel caso del Sole24Ore e del bot automatico @influbot sul social network Mastodon, che attraverso un report settimanale, aggiorna i propri lettori sull’andamento influenzale.
Lo scopo principale del progetto è accelerare il lavoro di ricercatori e funzionari della sanità pubblica per prepararsi, rispondere e ridurre il peso delle future emergenze sanitarie, colmando le criticità di raccolta dei dati durante le prime fasi di un’epidemia, come dimostrato con Ebola, e supportare le indagini epidemiologiche per risalire alla fonte dell’infezione, come fatto con le epatiti di origine sconosciuta nei bambini.
L’esperienza maturata negli ultimi anni ha permesso di integrare gli attuali flussi di dati con i recenti eventi epidemiologici riguardanti l’influenza aviaria che continua a diffondersi in tutto il mondo, uccidendo uccelli selvatici e pollame in massa, e causando sporadiche infezioni negli uomini. La morte di una bambina di 11 anni il 22 febbraio 2023 in Cambogia, a seguito del contatto con animali infetti, ha riacceso i riflettori sui rischi di una possibile nuova pandemia. Spostandosi lungo le rotte migratorie degli uccelli verso l'Europa e le Americhe, un nuovo ceppo di influenza H5N1 si è insediato negli uccelli selvatici e nel pollame domestico, portando a un record di 58,6 milioni di volatili abbattuti negli Stati Uniti e a nuovi focolai in America Latina e nei Caraibi. Lo scorso ottobre il virus si è probabilmente trasmesso tra visoni d'allevamento in Spagna e potrebbe essersi diffuso tra i leoni marini in Perù all'inizio di quest'anno.
Tutti questi episodi dimostrano come l’aviaria sia un agente patogeno altamente mutevole e soggetto a drastici cambiamenti genetici. Per sostenere gli sforzi di sorveglianza globale del virus è stata creata una line list ad hoc, organizzata come un foglio di calcolo con righe e colonne, per fornire informazioni affidabili e sintetizzate sui casi confermati in laboratorio e descritti nei rapporti ufficiali dell'OMS e da testate giornalistiche online attendibili (per esempio BNO News). Ogni riga rappresenta un caso e ogni colonna descrive una variabile epidemiologica chiave come: le date di insorgenza dei sintomi, di ospedalizzazione e di eventuale morte del paziente; le informazioni demografiche (età e sesso del paziente) e geografiche (nazione, provincia, città del paziente); qualsiasi informazione aggiuntiva in grado di descrivere con il maggior grado di accuratezza possibile l’infezione (sintomi, fonte di esposizione ed esito clinico del paziente). Inoltre, viene data la possibilità di consultare e usare i principali dati ufficiali sui focolai di influenza aviaria ad alta patogenicità (HPAI) in formato machine-readable, per consentire una rapida e coordinata pianificazione delle misure di prevenzione e controllo più adatte a ciascuna realtà geografica.
Le fonti ufficiali di notifica delle positività utilizzate per la creazione del database sono: il portale Avian Flu Data Portal, sviluppato dal Laboratorio europeo di riferimento sull’Influenza aviaria e la Malattia di Newcastle (EURL AI/ND) dell’Istituto Zooprofilattico Sperimentale delle Venezie (IZSVe); i rapporti trimestrali pubblicati congiuntamente dall’Autorità Europea per la Sicurezza Alimentare (EFSA)/ECDC/EURL; il sito web del dipartimento dell’Agricoltura degli Stati Uniti d’America (USDA); il sito istituzionale GOV.UK.
Un esempio di uso dei dati è mostrato in figura 2, che attraverso una visualizzazione interattiva Google Maps descrive i focolai segnalati in Italia da settembre 2021 al 20 aprile 2023.

Figura 1. Situazione epidemiologica dell’influenza aviaria in Italia per i virus ad alta patogenicità (HPAI). Fonte dati: https://www.izsvenezie.com/reference-laboratories/avian-influenza-newcas..., consultato il 20 aprile 2023
In futuro la piattaforma avrà l’obiettivo di migliorare le attività di routine (monitoraggio e valutazione del rischio epidemico, aggiunta di nuovi flussi di dati, ecc.) coinvolgendo direttamente esponenti delle autorità nazionali di salute pubblica e creando un feedback loop tra esperto e utente. È necessario che iniziative simili siano un esempio per sviluppare una politica lungimirante in grado di mettere i professionisti nelle condizioni di lavorare e fare analisi sui dati per provare a comprendere le dinamiche evolutive di una malattia. Occorre fare in modo che i diversi sistemi di sorveglianza non solo gestiscano i medesimi processi, ma lo facciano parlando la stessa lingua attraverso l’utilizzo di standard condivisi. La grave situazione di crisi emergenziale degli ultimi anni ha aumentato il senso di coesione e la percezione che i comportamenti altrui finiscono per influenzare la vita di ognuno di noi, per cui sarà fondamentale uno sforzo corale per il raggiungimento di obiettivi strategici condivisi.
Usare i dati per prevenire lo spillover in quattro mosse
Decenni di ricerche di epidemiologia, ecologia e genetica suggeriscono che una strategia globale efficace per ridurre il rischio di spillover dovrebbe concentrarsi su quattro azioni.
1 È necessario proteggere le foreste tropicali e subtropicali. Diversi studi dimostrano che i cambiamenti nelle modalità di utilizzo del territorio potrebbero essere il principale motore delle malattie infettive emergenti di origine zoonotica a livello globale. La fauna selvatica che sopravvive al disboscamento o al degrado delle foreste tende a includere specie che possono vivere accanto all'uomo e che spesso ospitano agenti patogeni in grado di infettare l'uomo. Per esempio, in Bangladesh, i pipistrelli portatori del virus Nipah, che può uccidere il 40-75% delle persone infettate, ora si trovano in aree ad alta densità di popolazione umana, perché il loro habitat forestale è stato quasi interamente disboscato. Tuttavia, le foreste possono essere protette anche mentre si aumenta la produttività agricola, a patto che ci siano la volontà politica e le risorse sufficienti. Lo dimostra la riduzione del 75% della deforestazione in Amazzonia nel periodo 2004-2012 (rispetto al decennio 1996-2005), grazie soprattutto a un migliore monitoraggio, all'applicazione delle leggi e alla concessione di incentivi finanziari agli agricoltori.
2 I mercati commerciali e il commercio di animali selvatici vivi che rappresentano un rischio per la salute pubblica devono essere vietati o strettamente regolamentati, sia a livello nazionale che internazionale. Ciò sarebbe coerente con l'appello lanciato dall'OMS e da altre organizzazioni nel 2021 affinché i paesi sospendano temporaneamente il commercio di mammiferi selvatici catturati vivi e chiudano le sezioni dei mercati che vendono tali animali. Diversi paesi hanno già agito in questo senso: in Cina, il commercio e il consumo della maggior parte della fauna selvatica terrestre sono stati vietati in risposta a COVID-19; analogamente, il Gabon ha vietato la vendita di alcune specie di mammiferi come cibo nei mercati.
3 È necessario migliorare la biosicurezza quando si tratta di animali da allevamento. Tra le altre misure, questo obiettivo potrebbe essere raggiunto attraverso una migliore assistenza veterinaria, una maggiore sorveglianza delle malattie animali, il miglioramento dell'alimentazione e degli alloggi degli animali e le quarantene per limitare la diffusione dei patogeni. Le cattive condizioni di salute degli animali d'allevamento aumentano il rischio di contrarre l'infezione da agenti patogeni e di diffonderli. Inoltre, quasi l'80% degli agenti patogeni del bestiame può infettare più specie ospiti, compresi gli animali selvatici e gli esseri umani.
4 Occorre migliorare la salute e la sicurezza economica delle persone. Le persone in cattive condizioni di salute, come quelle che soffrono di malnutrizione o di infezione da HIV non controllata, possono essere più suscettibili agli agenti patogeni zoonotici. Inoltre, alcune comunità, soprattutto quelle delle aree rurali, utilizzano le risorse naturali per produrre beni o generare reddito e questo li porta a entrare in contatto con la fauna selvatica o con i suoi derivati. In Bangladesh, per esempio, la linfa delle palme da dattero, consumata come bevanda in varie forme, viene estratta incidendo il tronco della palma, a cui viene fissato un contenitore per raccogliere il liquido che fuoriesce dall'incisione. Il contenitore può essere, però, contaminato con urina o saliva dai pipistrelli infetti. Un'indagine del 2016 ha collegato questa pratica a 14 infezioni da virus Nipah nell'essere umano, che hanno causato otto decessi.
È fondamentale fornire alle comunità opportunità educative insieme a servizi di assistenza sanitaria e di formazione su tecniche di sostentamento alternative, come l'agricoltura biologica. Per esempio, gli interventi progettati dall'organizzazione non governativa Health in Harmony nel periodo 2007-17 nel Borneo indonesiano, hanno contribuito a ridurre del 90% il numero di famiglie che dipendevano dal disboscamento illegale per il loro principale sostentamento. Questo, a sua volta, ha ridotto la perdita di foresta pluviale locale del 70%. Anche la mortalità infantile è diminuita del 67% nel bacino di utenza del programma educativo.
In termini di monitoraggio e risposta alle epidemie, COVID-19 ha evidenziato alcuni sviluppi promettenti nell'integrazione e nell'analisi dei dati, ma ha anche lasciato in eredità alcune sfide chiave.
Standardizzazione. L'acquisizione di dati epidemiologici provenienti da tutto il mondo presenta notevoli difficoltà, soprattutto a causa della diversità dei formati con cui i dati vengono raccolti, sintetizzati e diffusi. Pertanto, un passo fondamentale per facilitare la condivisione dei dati tra diversi paesi è quello di definire un formato standard che sia comune a tutti i diversi sistemi di reporting sanitario.
Qualità. La qualità dei dati può variare in modo sostanziale tra i diversi flussi di informazione, soprattutto nelle prime fasi di un'epidemia quando sono spesso meno strutturati. Pertanto, mentre i ricercatori devono valutare autonomamente la fattibilità di dati specifici per supportare i risultati dei loro studi, è indispensabile disporre di un modello decentralizzato, in cui volontari e membri di team di diverse regioni eseguono la convalida delle informazioni, come dimostrato dal COVID Tracking Project. Per le epidemie future, sarà importante dunque automatizzare i processi di convalida dei dati attraverso finanziamenti stabili e approcci flessibili, perché ogni focolaio è diverso dall'altro. Inoltre, poiché tutti i focolai di malattie infettive rischiano di diventare globali, un database completo a livello mondiale sarà utile per guidare risposte coordinate.
Sostenibilità. Il volume e la diversità delle informazioni generate durante la pandemia COVID-19 sono stati senza precedenti. Per elaborare e analizzare questi insiemi di dati, gli scienziati hanno implementato nuovi strumenti e aggiornato software esistenti. Tuttavia, l’emergenza sanitaria ha messo in evidenza la necessità di avere una visione più ampia dello sviluppo e della manutenzione delle risorse, poiché i dati e i software open source di successo necessitano di un continuo monitoraggio per rispondere rapidamente alle mutevoli esigenze delle loro comunità di utenti. In futuro, la collaborazione di partner diversi come università, organizzazioni sanitarie e gruppi del settore privato potrebbe consentire lo sviluppo di una rete multidisciplinare e multisettoriale, capace di coinvolgere in un’ottica “One Health” non solo medici ma anche le altre figure professionali che possano contribuire alle attività di prevenzione, previsione, rilevazione e risposta alle minacce sanitarie globali.
Equità. I recenti eventi storici hanno ricordato al mondo quanto sia fragile e al tempo stesso interconnesso. Ciò che minaccia alcuni può presto minacciare l’intera comunità. La raccolta, l'integrazione e la diffusione di dati tempestivi dovranno seguire delle linee guida che impediscano l'utilizzo delle informazioni per rafforzare i pregiudizi esistenti o la discriminazione nei confronti di popolazioni specifiche in base al sesso, all'età o all'ubicazione. È inoltre fondamentale che in futuro, dati e codice software siano open source per consentire una rapida integrazione tra più gruppi di ricerca e governi nazionali. Una piattaforma veramente aperta può aiutare gli utenti a superare le barriere geografiche, organizzative e sociali esistenti per l'accesso alle informazioni e consentire una grande responsabilizzazione e democratizzazione della salute pubblica.